Neuer Ansatz zur CRISPR-Regulierung
Ein Protein inhibiert bestimmte CRISPR-Genscheren auf völlig neue Weise. Der nun aufgeklärte Mechanismus bietet einen neuen Ansatz zur gezielten Steuerung von CRISPR-Technologien.
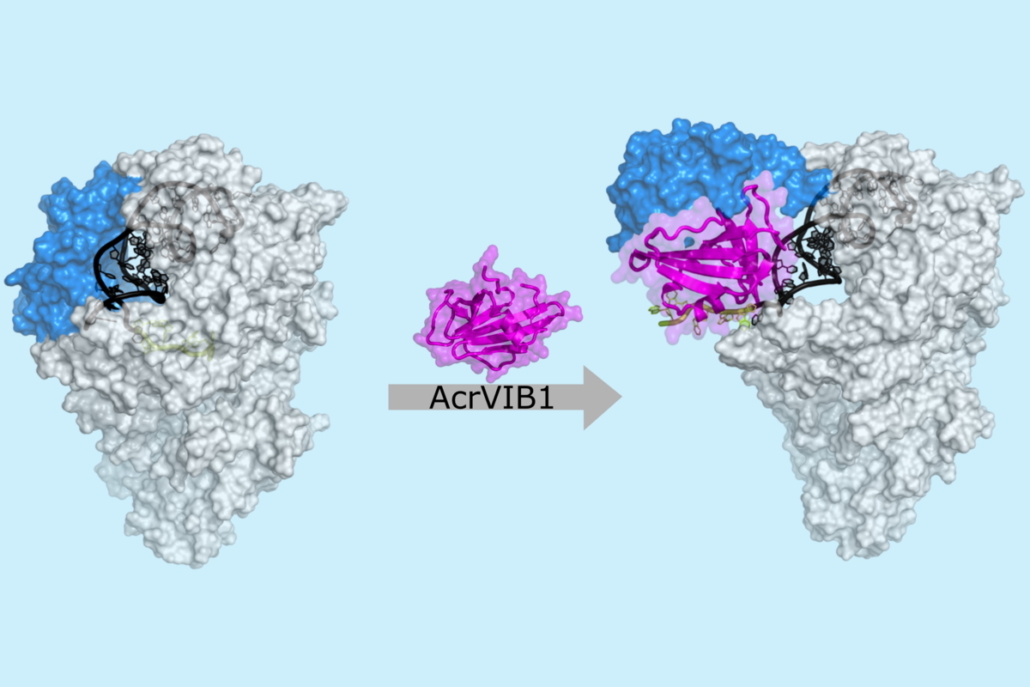
Die CRISPR-Cas-Genschere bietet vielseitige Einsatzmöglichkeiten, unter anderem in der Diagnostik, für die Therapie genetischer Erkrankungen oder viraler Infektionen. Eine gezielte Stilllegung der Genschere durch Protein-Inhibitoren ist unerlässlich, um ihre Funktion feinregulieren und die Sicherheit ihrer Anwendung gewährleisten zu können. Eine große Familie solcher Anti-CRISPR-Proteine ist bereits bekannt. Nun konnte ein Forschungsteam den ungewöhnlichen Mechanismus von AcrVIB1 aufklären, der sich gegen die Cas13b-Nuklease richtet. Statt den Aufbau des CRISPR-Komplexes zu stören, hemmt AcrVIB1dessen Funktion durch feste Bindung der Nuklease, wie im Fachjournal Molecular Cell beschrieben ist.
Das Team um Wulf Blankenfeldt vom Braunschweiger Helmholtz-Zentrum für Infektionsforschung (HZI) und Chase L. Beisel vom Würzburger Standort, dem Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI), arbeitet daran, neue Acr-Proteine zu identifizieren. So entdeckten die Wissenschaftler auch AcrVIB1 als erstes Anti-CRISPR-Protein gegen die Cas13b-Nuklease. Im Gegensatz zu Cas9 oder Cas12-Proteinen richten sich Cas13-Proteine vom Typ VI, zu denen auch der Untertyp Cas13b gehört, gezielt gegen RNA-Moleküle und werden daher für RNA-Interferenz oder antivirale Anwendungen eingesetzt. Hierfür benötigt das Cas-Protein eine CRISPR-Leit-RNA (crRNA), um die komplementäre RNA von Phagen zu erkennen. Die Nuklease kann die Ziel-RNA und auch andere RNAs in der Nähe schneiden, woraufhin diese RNAs abgebaut werden. Die Vielfalt wird deutlich durch ein weiteres Nuklease-System, das erst kürzlich beschrieben wurde: Das flexible und besonders kompakte TIGR-Tas-System arbeitet stattdessen mit tigRNAs (Tandem Interspaced Guide RNAs) und kann mit beiden DNA-Strängen ohne die Notwendigkeit bestimmter Motive interagieren, um so zielgenau jede Stelle des Genoms zu verändern.
Anti-CRISPR-Proteine sind eine der größten Gruppen von bekannten Inhibitoren. Mittlerweile sind über 100 verschiedene Acr-Proteine beschrieben. Zwischen Bakterien und Viren hat sich ein regelrechtes evolutionäres Wettrüsten entwickelt. Virale Acr-Proteine hemmen verschiedene Typen von CRISPR-Cas-Abwehrsystemen auf ganz unterschiedliche Weise. Bakterien wiederum umgehen die virale Infektion durch Erkennung von Acr-Proteinen. In den meisten Fällen wird die Erkennung der Ziel-RNA oder die Bindung der crRNA verhindert.
AcrVIB1 hingegen verstärkt diese Bindung sogar. Der Komplex ist jedoch funktionsunfähig. Den genauen Hemmmechanismus konnte das Team in der aktuellen Studie mittels hochauflösender Bildgebung durch Kryo-Elektronenmikroskopie aufklären. „Die engere Bindung zwischen Nuklease und Leit-RNA war völlig unerwartet. Ein einfacherer und deswegen von uns zuerst erwarteter Mechanismus hätte darin bestanden, die Kopplung der Leit-RNA von vornherein zu verhindern“, so die Autoren. Der von AcrVIB1 eingeschlagene Weg sei jedoch sogar effektiver: AcrVIB1 bindet sich fest an Cas13b und inaktiviert so die Nuklease. Gleichzeitig bleibt die crRNA-Bindungsregion frei, was den Abbau der Leit-RNAs durch zelluläre Ribonukleasen beschleunigt. Cas13b werde zu einer Art Sackgasse für crRNAs.
“Unsere Erkenntnis liefert eine Blaupause für die Entwicklung von Molekülen, die die Funktion des Anti-CRISPR-Proteins nachahmen oder verändern könnten“, sagte Seniorautor Blankenfeldt mit Blick auf mögliche Anwendungen. „Wir könnten künftig Moleküle wie AcrVIB1 gezielt einsetzen, um CRISPR-Systeme in verschiedenen Anwendungen zu regulieren oder vorübergehend zu deaktivieren.“ Neben einem tieferen Verständnis der viralen Anti-CRISPR-Strategien, sollen die Erkenntnisse auch zu innovativen Therapien und Diagnoseverfahren in der Medizin beitragen. So könnten sie die Präzision von RNA-Tests wie SHERLOCK und DETECTR verbessern.

 Leibniz-Institut für Pflanzengenetik und Kulturpflanzenforschung IPK Gatersleben, Andreas Baehring
Leibniz-Institut für Pflanzengenetik und Kulturpflanzenforschung IPK Gatersleben, Andreas Baehring Robert Hoffie
Robert Hoffie Screenshot Biocom interrelations GmbH
Screenshot Biocom interrelations GmbH